肺癌是全球癌癥致死的首位原因,而小細胞肺癌約占肺癌總數的15%,是所有肺癌亞型中惡性程度最高、預后最差的亞型,其5年生存率僅為5%。與非小細胞肺癌形成鮮明對比,小細胞肺癌的治療手段單一,患者總生存率止步不前。分子表征與組學研究的不足極大限制了小細胞肺癌的基礎和臨床進展,迄今為止,僅有少量針對小細胞肺癌臨床樣本的基因組研究被報道,又由于小細胞肺癌普遍存在TP53和RB1的失活突變,缺乏驅動突變,使得難以通過基因突變信息獲得有效靶點和分子分型。而蛋白質作為生命功能的執行者,是95%以上藥物的作用靶點,目前尚缺乏從蛋白質組層面對小細胞肺癌進行系統研究,因此,全面系統地表征小細胞肺癌的蛋白質組學圖譜,有利于深入理解小細胞肺癌的病理機制,對于實現更精準的小細胞肺癌分子分型和個性化治療具有重要意義。
該研究對小細胞肺癌患者的腫瘤組織和配對癌旁組織樣本進行了蛋白基因組學(proteogenomics)分析,通過整合基因組、轉錄組、蛋白質組、磷酸化蛋白質組等多維組學數據,系統揭示了小細胞肺癌的分子特征,提出新的分子分型及其針對性的個體化治療策略,為深入理解小細胞肺癌的發病機制和改善臨床治療策略奠定了堅實的基礎。
研究人員通過對蛋白質組學數據進行監督分析篩選預后相關生物標志物,發現HMGB3的高表達與患者不良預后密切相關,并通過免疫組化實驗在獨立隊列臨床樣本中得以驗證。生物學實驗表明,HMGB3可通過轉錄調控細胞連接 (cell-junction) 相關基因表達促進小細胞肺癌的細胞遷移。
其次,研究人員系統分析了小細胞肺癌的免疫微環境特征,發現ZFHX3突變與免疫細胞浸潤水平升高密切相關。更重要的是,在接受PD-1或PD-L1抑制劑聯合化療的小細胞肺癌臨床試驗患者腫瘤樣本中,研究人員證實了ZFHX3突變患者呈現更好的治療響應,提示ZFHX3突變或可作為小細胞肺癌免疫治療受益的生物標志物。
最后,研究人員利用多組學數據基于非負矩陣分解算法將小細胞肺癌分為四個亞型(nmf1-4),系統表征了各亞型獨特的分子特征并提出潛在治療策略。其中,nmf1為經典亞型,表現為增殖快、E2F活性高、復制壓力高,提示可能受益于基于順鉑+依托泊苷的一線化療方案;nmf2亞型腫瘤表現出顯著升高的DLL3表達,提示可能受益于靶向DLL3的治療方案;nmf3亞型腫瘤在磷酸化蛋白質組層面表現出受體酪氨酸激酶(RTK)通路的顯著激活,提示可能受益于靶向RTK的治療方案;nmf4亞型患者表現出特異性升高的MYC表達及MYC通路激活,提示可能受益于極光激酶(AURK)抑制劑。研究人員還建立了大規模的小細胞肺癌人源腫瘤異種移植(PDX)模型和細胞系移植瘤(CDX)模型,并對其進行基因組學和蛋白質組學整合分析,在進行類似分子分型的基礎上驗證了基于臨床樣本分子分型的針對性治療策略。
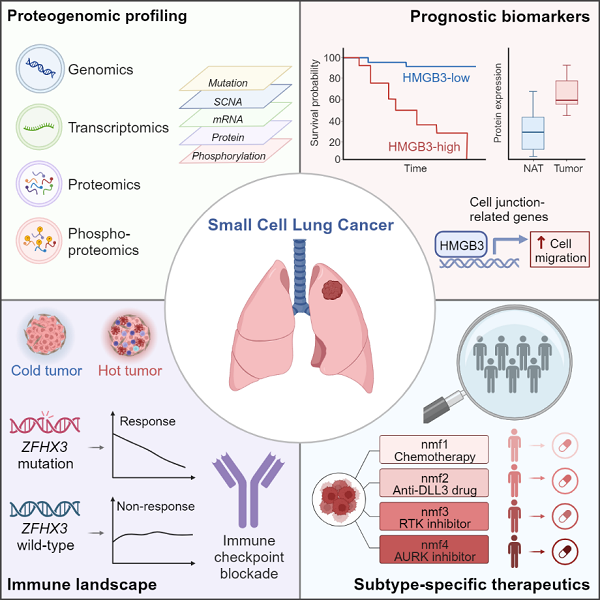
圖:小細胞肺癌蛋白基因組學分析
該研究是國際上首次大規模對小細胞肺癌臨床隊列開展蛋白質組和磷酸化蛋白質組表征,這些研究成果為小細胞肺癌的病理機制解析、預后檢測、分子分型及個性化治療提供了理論依據,同時產生的高質量大數據將為廣大小細胞肺癌的基礎與臨床研究者提供支持,推動小細胞肺癌研究領域的發展。
近年來,周虎課題組通過開展合作,采用以蛋白質組學為核心的多組學技術,在臨床腫瘤蛋白質組學研究等方面取得了一系列科研成果(Cell,2019;Cancer Cell,2022; National Science Review, 2023),為腫瘤的發生發展機制和潛在精準化靶向治療方案提供了理論基礎,為相關的基礎與臨床研究提供了重要支持。
張鵬教授、周虎研究員、季紅斌研究員和高大明研究員為該論文共同通訊作者。同濟大學附屬上海市肺科醫院/上海藥物所劉倩博士、上海市肺科醫院章靖博士、中國科學院分子細胞科學卓越創新中心郭晨晨博士、博士研究生王夢成、同濟大學生命科學與技術學院王晨飛教授為該論文共同第一作者。該工作得到了同濟大學博士研究生孫良棟、嚴以律,上海市肺科醫院張樂樂副研究員、美國國立癌癥研究院Henry Rodriguez博士、Ana I. Robles博士、華盛頓大學丁麗教授、美國貝勒醫學院章冰教授、約翰斯-霍普金斯大學張會教授、耶魯大學劉延盛教授的大力支持。該研究也是依托于上海藥物所公共技術中心的重大科研產出。研究獲得了國家自然科學基金委杰出青年科學基金項目、重大項目、科技部國家重點研發計劃、上海市優秀學術帶頭人項目、上海市市級科技重大專項、博士后科學基金等項目的資助。
原文鏈接:https://www.cell.com/cell/fulltext/S0092-8674(23)01335-1
參考文獻
1. Rudin CM, et al. Small-cell lung cancer. Nat Rev Dis Primers. 2021;7(1):3.
2. George J, et al. Comprehensive genomic profiles of small cell lung cancer. Nature. 2015;524(7563):47-53.
3. Gao Q, et al. Integrated Proteogenomic Characterization of HBV-Related Hepatocellular Carcinoma. Cell. 2019;179(2):561-577.e22.
4. Dong L, et al. Proteogenomic characterization identifies clinically relevant subgroups of intrahepatic cholangiocarcinoma. Cancer Cell. 2022;40(1):70-87.e15.
5. Zhu H, et al. Proteomics of adjacent-to-tumor samples uncovers clinically relevant biological events in hepatocellular carcinoma. Natl Sci Rev. 2023;10(8):nwad167.
(供稿部門:周虎課題組;供稿人:劉倩)




 17312606166
17312606166