SNP 在人類基因組中的發生頻率比較高,大約平均每1000個堿基對中就有一個多態位點,估計其總數可達300萬個甚至更多[2]。當SNP發生在基因編碼區或基因的調節區域時,它會對基因表達產生巨大的影響,從而影響基因的功能。有一些SNP位點已被證明在人類健康研究中非常重要,例如鐮狀細胞貧血、β地中海貧血和囊性纖維化的發生主要是由于SNP的出現。同時,研究人員發現,SNP可能有助于預測個體對某些藥物的反應、對毒素等環境因素的易感性以及罹患特定疾病的風險。例如,攜帶有APOE(載脂蛋白)ε4突變的個體表現出患阿爾茨海默病的更高風險[3,4](圖3)。

圖3 APOE4促進病理性tau蛋白的再分配。
a:人源APOE敲入小鼠海馬體tau蛋白染色結果;b:Tau蛋白陽性區域占海馬體齒狀回部位的面積 (TE2:APOE2敲入小鼠, TE3: APOE3敲入小鼠, TE4: APOE4敲入小鼠, TEKO: ApoE敲除小鼠)[4]
隨著測序技術的發展,全基因組關聯分析(Genome-wide Association Study,GWAS)已廣泛應用于復雜疾病的遺傳因素分析(圖4),可以更高效地獲得疾病與SNP之間的相關性。GWAS利用高通量測序技術,分析數以萬計的SNPs以及這些SNPs與臨床表型和可測性狀的相關性,從而全面地揭示出不同復雜性狀的遺傳機制。目前,全世界范圍內已開展了4000多項GWAS研究,發現了超過10萬個與各種疾病(如癌癥,高血壓,II型糖尿病,類風濕性關節炎等)以及重要生理性狀關聯的基因位點。

圖4 GWAS分析人群中等位基因的變異頻率(https://www.ebi.ac.uk/)
完成了GWAS分析之后,我們還需要借助下游工具對GWAS的分析結果進一步注釋和挖掘,篩選可能致病的SNP位點。相關的注釋網站和數據庫很多,以下推薦2個網站,供大家參考。GWAS4D是通過組織/細胞類型特異性和非編碼調控變異的優先排序,來預測特定復雜疾病和性狀的潛在致病機制。我們將GWAS summary數據導入,便可以輸出SNP位點的優先排序、SNP位點在基因組區域的可視化以及SNP功能預測和注釋等結果(圖7)。網址:http://mulinlab.tmu.edu.cn/gwas4d 。

圖5 GWAS4D可進行分析的界面
SNPedia是一個SNP百科全書類網站,它引用已經發布的文章或者數據庫信息,對SNP位點進行描述,共享著人類基因組變異的信息。我們可以搜索某個SNP位點來尋找與之相關的信息,也可以根據相關疾病和癥狀來尋找相關的SNP(圖6)。網址:https://www.snpedia.com/index.php/SNPedia。
圖6 SNPedia首頁
受益于二代測序及GWAS的聯合應用,研究人員發現了眾多和臨床表型具有一定聯系的SNP位點,然而要驗證這些位點與臨床表現的關系,必須回到體內進行研究。如果這些突變可以在小鼠體內找到,那么突變小鼠就能為臨床研究提供寶貴的信息。因此構建突變小鼠,是有效驗證臨床檢測結果的關鍵一步。
構建突變小鼠時,首先需要確定的是突變位點的人鼠同源性及保守性。小鼠是解析人類基因功能、研究人類疾病重要的模式生物。99%的人類基因都可以在其基因組中能找到相應的基因。但需要注意的是,人鼠編碼基因序列相似性平均為85%,而非編碼基因的相似度只有50%[5],因此找到同源基因還需要判斷突變位點的序列保守性。
這里舉一個例子,PLD3的一個SNP(rs145999145)是遲發性阿爾茲海默癥的危險因素之一。我們在NCBI-SNP數據庫可搜索到該SNP的位點信息,如下:
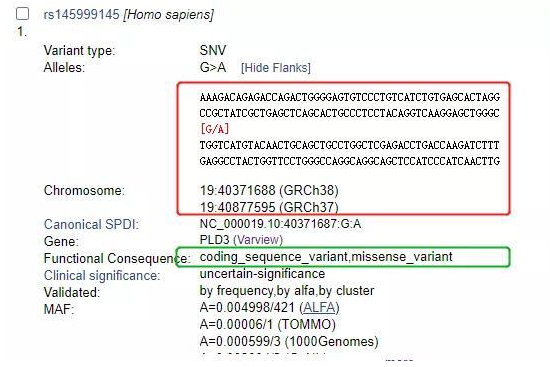
圖7 rs145999145在NCBI數據庫中的信息頁
可見紅色框中提供了突變位點的序列信息,而綠色框中提示了突變類型是錯義突變(圖7)。根據該位點附近的側翼序列以及給到的基因組位點,我們可以找到其在人源PLD3基因上的位置,轉錄本NM_012268.4,c.694 G>A,也即p.232V>M,位于exon9。通過對人鼠同源基因的同源性分析(圖8),可以很快找到相應小鼠的突變位點:NM_001317355.1,c.688 G>A,p.230V>M,位于exon10。

圖8 人鼠PLD3蛋白同源性對比結果
確定了小鼠的突變位點后,我們便可以針對該突變位點設計Guide RNA,利用Crispr/Cas9技術來構建點突變小鼠(圖9)。

圖9 PLD3-V230M突變小鼠構建策略
接下來和大家分享一個案例來具體說明如何利用基因修飾小鼠驗證SNP的臨床表型。
【標題】自身免疫疾病相關IgG1變體調控B淋巴細胞活化和分化的新機制
【期刊】Science (IF=41)

系統性紅斑狼瘡SLE是最常見的自身免疫疾病之一,是一種由遺傳、環境、免疫等多種因素導致的復雜性疾病。其發病機制至今尚不清楚,導致治療手段有限且無法根治。人體中廣泛存在具有自身反應性的IgG型記憶性B細胞,SLE等自身免疫疾病常伴隨大量自身抗體的產生。
研究人員猜想IgG型B細胞受體(IgG-BCR)的異常活化可能參與到SLE的發病過程。通過對患者進行基因檢測,研究人員發現,IgG1重鏈基因IGHG1上的SNP(rs117518546)在SLE患者中顯著增加,該SNP導致IgG1第396位甘氨酸突變為精氨酸(IgG1-G396R)(圖9)。攜帶該SNP的SLE患者產生更多更廣泛的IgG1型的自身抗體,發生炎癥反應的風險增加,疾病活動指數也更高,揭示該SNP為新的SLE易感基因位點。
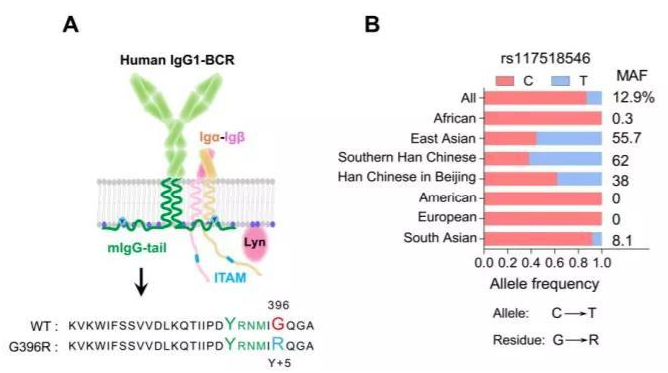
圖10 rs117518546突變位點(圖A)及在不同地區人群中的比例(圖B)
值得注意的是,該SNP在東亞人群中的出現的頻率遠高于歐美人群(圖10),這提示該SNP是以漢族為代表的東亞人群特有的SLE易感基因位點,可能是我國SLE患者與其他人群患者的臨床表現和治療反應存在差異的因素之一。
通過同源性對比,研究者發現該SNP在小鼠上的位點為390位甘氨酸突變為精氨酸,因此構建了IgG1-G390R突變小鼠(圖11)。通過誘導野生型小鼠和突變小鼠發生自身免疫疾病,研究者發現攜帶該SNP的小鼠產生更多、更廣泛的自身抗體,而這依賴于B細胞受抗原刺激后大量自身反應性漿細胞的分化(圖12)。這一結果驗證了在SLE患者中的發現,并從B細胞命運決定層面給出了初步解釋[6]。

圖11 構建IgG1-G390R敲入小鼠

圖12誘導自身免疫病發生后,野生型小鼠和G390R小鼠體內dsDNA和SmD抗體動態變化。
該研究從疾病易感基因位點的臨床遺傳學研究出發,經過基于基因修飾小鼠的動物模型研究,將臨床上發現的問題在真實的體內實驗中驗證,確認了IgG1-G396R在SLE疾病發生發展中的作用。這項研究為SLE研究提供了全新的研究靶點,也為復雜疾病相關易感基因位點的研究提供了創新性的研究模式,最終發表在《Science》雜志上。
南模生物深耕基因修飾動物模型行業二十余年,在構建類人SNP同源突變小鼠方面具有豐富的經驗,助力客戶發表在《Cell Research》、《Immunity》等多種高水平雜志上,受到廣泛認可。歡迎大家咨詢,快來定制專屬您的點突變小鼠吧。
參考文獻:
[1]楊煥明. 人類基因組計劃及其意義——規模化、序列化、信息化與產業化、醫學化、人文化[J]. 科技與法律, 2000(3):2.
[2] Altshuler D, Pollara VJ, Cowles CR. An SNP map of the human genome generated by reduced representation shotgun sequencing. Nature. 2000 Sep 28;407(6803):513-6.
[3] https://www.snpedia.com/index.php/APOE-%CE%B54
[4] Shi Y, Yamada K, Liddelow SA, et al. ApoE4 markedly exacerbates tau-mediated neurodegeneration in a mouse model of tauopathy. Nature. 2017 Sep 28;549(7673):523-527. doi: 10.1038/nature24016
[5] https://www.nih.gov/news-events/news-releases/new-comprehensive-view-mouse-genome-finds-many-similarities-striking-differences-human-genome
[6] Chen X, Sun X, Yang W, et al. An autoimmune disease variant of IgG1 modulates B cell activation and differentiation. Science. 2018 Nov 9;362(6415):700-705. doi: 10.1126/science.aap9310.




 17312606166
17312606166